Dans cet article, j’utiliserai un ensemble de données sur le Corona Virus, pour effectuer une analyse de données sur le Covid-19 avec Python, NumPy et Pandas (statistiques sur tous les pays affectés par le Covid-19).
Le dataset utilisé est disponible sur Kaggle : https://www.kaggle.com/sudalairajkumar/novel-corona-virus-2019-dataset.
Pour cette analyse de données, j’ai utilisé Python, NumPy et Pandas. Et j’ai utilisé l’outil Google Colab pour le code, vous le trouverez en cliquant ici.
Les données sur le Covid-19
Cet ensemble de données est composé de plusieurs fichiers :
- covid_19_data.csv – contient une liste d’observations pour tous les différents Etats/provinces de chaque pays. Il contient des observations pour les cas confirmés, les décès et les cas de rétablissement.
- COVID19_line_list_data.csv – contient une liste détaillée de cas avec des détails tels que le lieu, les symptômes, les visites à l’hôpital, ainsi que la source d’information, etc.
- COVID19_open_line_list.csv – similaire au fichier précédent (COVID19_line_list_data.csv).
- time_series_covid_19_confirmed_US – Une ventilation détaillée de tous les cas confirmés aux États-Unis. Contient des détails tels que le lieu, la ville et l’État.
- time_series_covid_19_deaths_US.csv – Une ventilation détaillée de tous les cas de décès aux États-Unis. Contient des détails tels que le lieu, la ville et l’état.
- time_series_covid_19_confirmed.csv – Contient la liste de tous les cas confirmés pour tous les pays du monde. Il énumère les provinces/états pour chaque pays, ainsi que la géolocalisation de chaque lieu.
- time_series_covid_19_deaths.csv – Contient la liste de tous les cas de décès pour tous les pays du monde. Il énumère les provinces/états pour chaque pays, ainsi que la géolocalisation de chaque lieu.
- time_series_covid_19_recovered.csv – Contient la liste de tous les cas retrouvés pour tous les pays du monde. Il énumère les provinces/états pour chaque pays, ainsi que la géolocalisation de chaque lieu.
Pour cet article, je me suis intéressé à l’exploration des différentes statistiques pour chaque pays touché par Covid-19. Ainsi, je me suis particulièrement intéressé à ces trois fichiers CSV :
- time_series_covid_19_confirmed.csv
- time_series_covid_19_deaths.csv
- time_series_covid_19_recovered.csv
En fait, j’aurai pu aussi utiliser le fichier covid_19_data.csv, car il regroupe en un seul fichier tous les cas confirmés, ceux qui sont rétablis et les décès. Malheureusement, il ne contient pas la latitude et la longitude de chaque lieu (que j’aimerais utiliser pour tracer sur une carte). J’ai donc décidé d’utiliser les trois fichiers CSV distincts.
À ce stade, il est bon que vous ouvriez les fichiers CVS ci-dessus à l’aide d’Excel et que vous exploriez les différentes colonnes de chaque fichier.
Analyse de données sur le Covid-19
Importation des bibliothèques
La première chose à faire pour tous les projets d’analyse de données est d’importer les modules et paquets pertinents :
import pandas as pd import numpy as np
Lecture des données
Pour cet article, l’ensemble de données que j’ai utilisé est daté du 14 juin 2020.
Pour votre propre expérimentation, je vous recommande de télécharger le dernier ensemble de données et de suivre l’analyse suivante.
J’ai donc enregistré les fichiers CSV dans un dossier nommé “Dataset – covid – 14 juin 2020”.
Chargeons dès maintenant les fichiers CSV dans des DataFrames Pandas :
dataset_path = "./Dataset - covid - 14 juin 2020/"
df_conf = pd.read_csv(dataset_path +
"time_series_covid_19_confirmed.csv")
df_death = pd.read_csv(dataset_path +
"time_series_covid_19_deaths.csv")
df_recovered = pd.read_csv(dataset_path +
"time_series_covid_19_recovered.csv")
print(df_conf.shape)
print(df_death.shape)
print(df_recovered.shape)
Observez la forme de chaque DataFrame :
(266, 129) (266, 129) (253, 129)
À ce stade, notez que le DataFrame df_recovered a une forme différente des deux autres. Essayons de creuser davantage et de voir pourquoi il a moins de lignes que les deux autres.
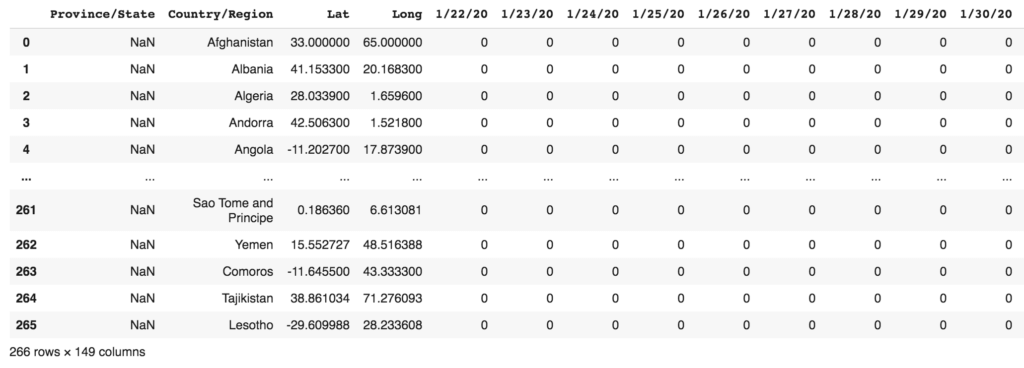
Voyons voir à quoi ressemble le DataFrame df_conf :
df_conf
On obtient ceci :
Tri et observation des DataFrames
Trions maintenant les DataFrames par Province/State et Country/Region:
df_conf = df_conf.sort_values(
by=['Province/State','Country/Region'])
df_conf = df_conf.reset_index(drop=True)
df_death = df_death.sort_values(
by=['Province/State','Country/Region'])
df_death = df_death.reset_index(drop=True)
df_recovered = df_recovered.sort_values(
by=['Province/State','Country/Region'])
df_recovered = df_recovered.reset_index(drop=True)
Voyons rapidement à quoi ressemblent les DataFrames.
df_conf
Vous obtiendrez le résultat suivant :
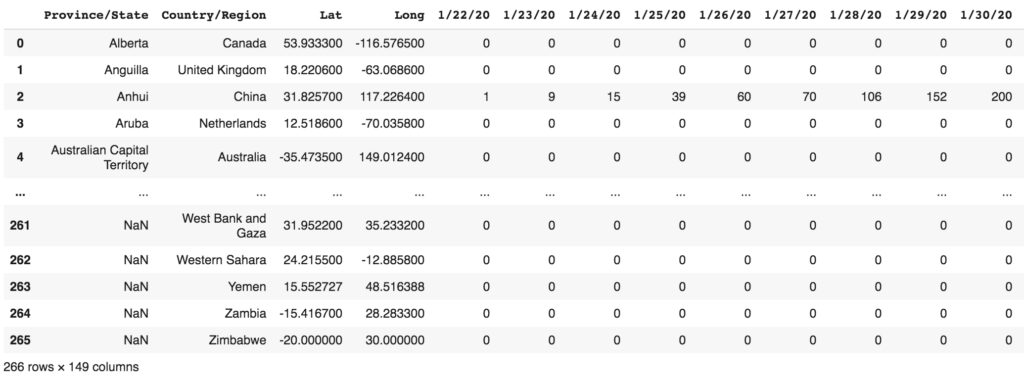
Les deux autres DataFrames (df_death et df_recovered) sont similaires. Observez ce qui suit : les dates de chaque cas signalé sont stockées dans des colonnes. À partir de la colonne 4, elle répertorie le cas signalé pour chaque jour – 1/22/2020, 1/23/2020, etc.
Pour notre analyse, nous allons devoir convertir toutes ces dates en une seule colonne, une autre colonne représentant les chiffres réels pour chaque jour. Mais avant cela, comparons les trois DataFrames pour voir s’ils contiennent les mêmes lignes :
# Vérifie si des colonnes de 2 dataframes sont les mêmes
print(df_conf[["Province/State","Country/Region"]].equals(
df_death[["Province/State","Country/Region"]]))
'''
True
'''
print(df_conf[["Province/State","Country/Region"]].equals(
df_recovered[["Province/State","Country/Region"]]))
'''
False
'''
Comme vous pouvez le voir dans les résultats ci-dessus, les DataFrames df_conf et df_death contiennent les mêmes éléments pour les deux colonnes – Province/State et Country/Region. Toutefois, elles sont différentes du DataFrame df_recovered.
La raison, c’est que le fichier time_series_covid_19_recovered.csv enregistre le Canada comme un seul pays alors que les deux autres fichiers CSV enregistrent le Canada avec ses différents États (individuellement) :
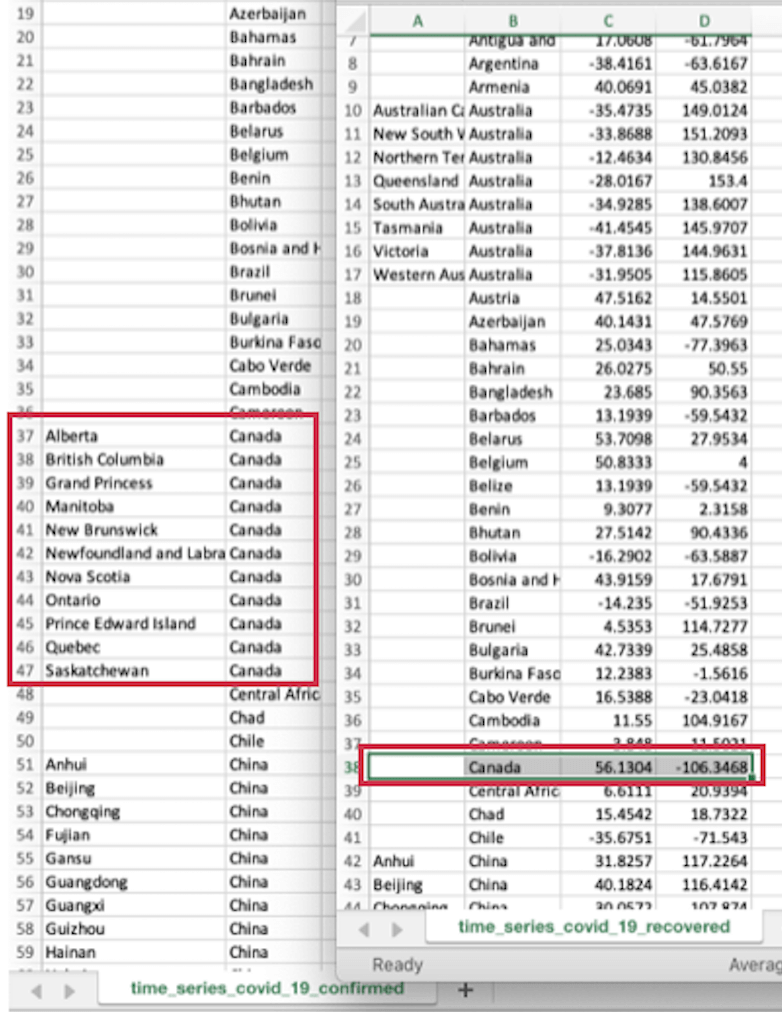
Pour cette raison (et pour garder les choses relativement simples pour cet article), je vais omettre le jeu de données pour les cas de rétablissement (time_series_covid_19_recovered.csv).
Extraire les noms des colonnes contenant des dates
Nous allons maintenant extraire les différentes dates qui sont enregistrées dans les fichiers CSV :
dates_conf = df_conf.columns[4:]
dates_death = df_death.columns[4:]
print(dates_conf)
print(dates_death)
# s'assurer que les dates sont les mêmes pour les 2 DataFrames
if dates_conf.equals(dates_death):
print("Les dates sont les mêmes")
On obtient en sortie le résultat suivant :
Index(['1/22/20', '1/23/20', '1/24/20', '1/25/20', '1/26/20', '1/27/20',
'1/28/20', '1/29/20', '1/30/20', '1/31/20',
...
'6/5/20', '6/6/20', '6/7/20', '6/8/20', '6/9/20', '6/10/20', '6/11/20',
'6/12/20', '6/13/20', '6/14/20'],
dtype='object', length=145)
Index(['1/22/20', '1/23/20', '1/24/20', '1/25/20', '1/26/20', '1/27/20',
'1/28/20', '1/29/20', '1/30/20', '1/31/20',
...
'6/5/20', '6/6/20', '6/7/20', '6/8/20', '6/9/20', '6/10/20', '6/11/20',
'6/12/20', '6/13/20', '6/14/20'],
dtype='object', length=145)
Les dates sont les mêmes
Afficher nos DataFrames dans un format contenant une colonne pour les dates, une pour les cas confirmés et une autre pour les décès
Je veux maintenant “supprimer le pivot (unpivot)” des DataFrames afin que les dates ne soient plus représentées sous forme de colonnes. Les dates doivent plutôt être stockées en tant que valeurs dans une nouvelle colonne, par exemple la colonne Date.
Le nombre de cas (Confirmed, Deaths) doit être enregistré dans une colonne correspondante, par exemple les colonnes Confirmed et Deaths. Pour cela, je peux utiliser la méthode melt() :
df_conf_melted = df_conf.melt(id_vars=['Province/State',
'Country/Region', 'Lat', 'Long'],
value_vars=dates_conf,
var_name='Date',
value_name='Confirmed')
df_death_melted = df_death.melt(id_vars=['Province/State',
'Country/Region', 'Lat', 'Long'],
value_vars=dates_death,
var_name='Date',
value_name='Deaths')
Voir les Dataframes résultants
Vous pouvez maintenant visualiser les DataFrames auxquels on a en quelque sorte retiré les pivots.
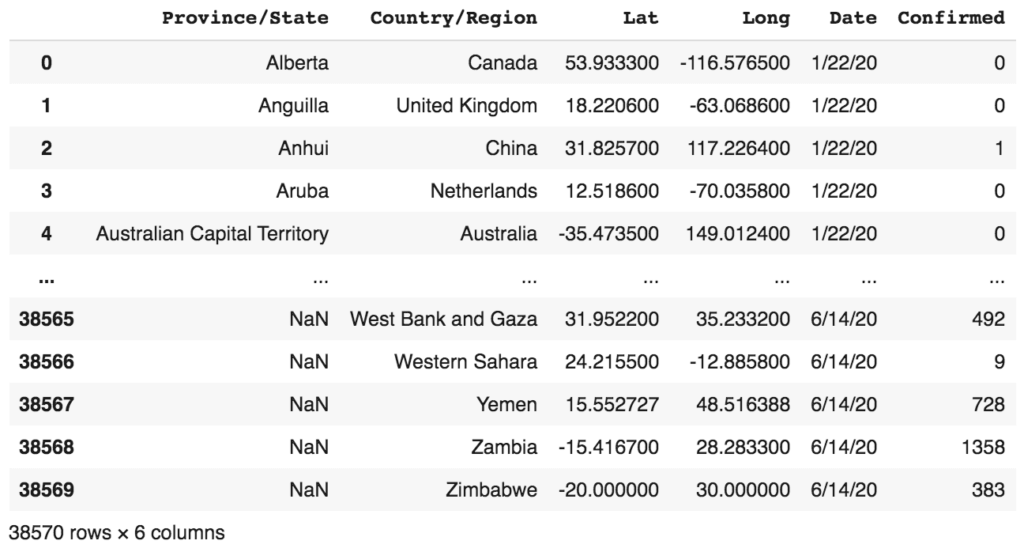
Commençons par df_conf_melted :
df_conf_melted
On obtient :
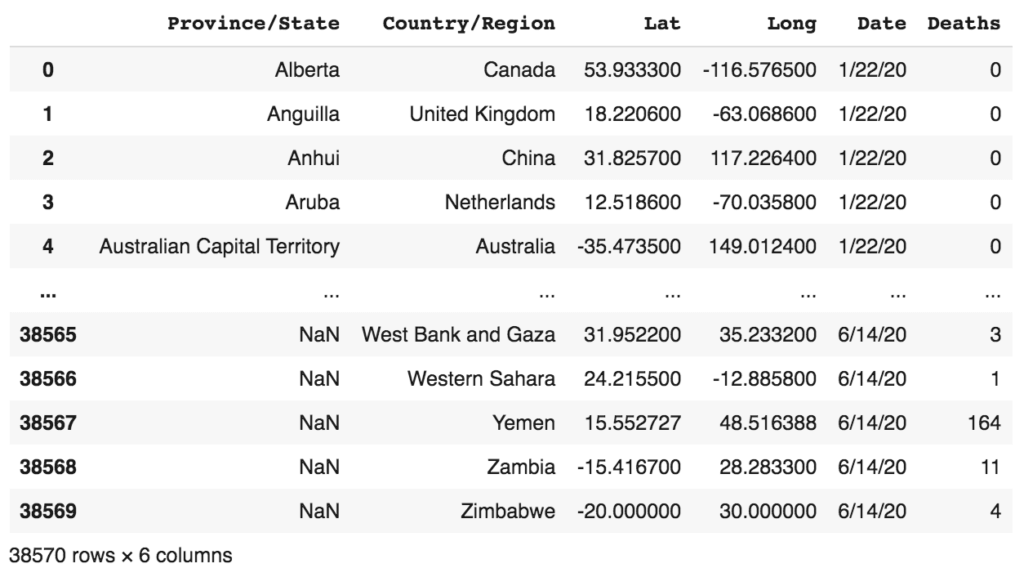
Regardons aussi le DataFrame df_death_melted :
df_death_melted
Ce qui donne :
Juste pour s’assurer que les DataFrames ont toujours la même forme :
print(df_conf_melted.shape) print(df_death_melted.shape) ''' (38570, 6) (38570, 6) '''
Combiner tous les 2 Dataframes résultants en un seul DataFrame
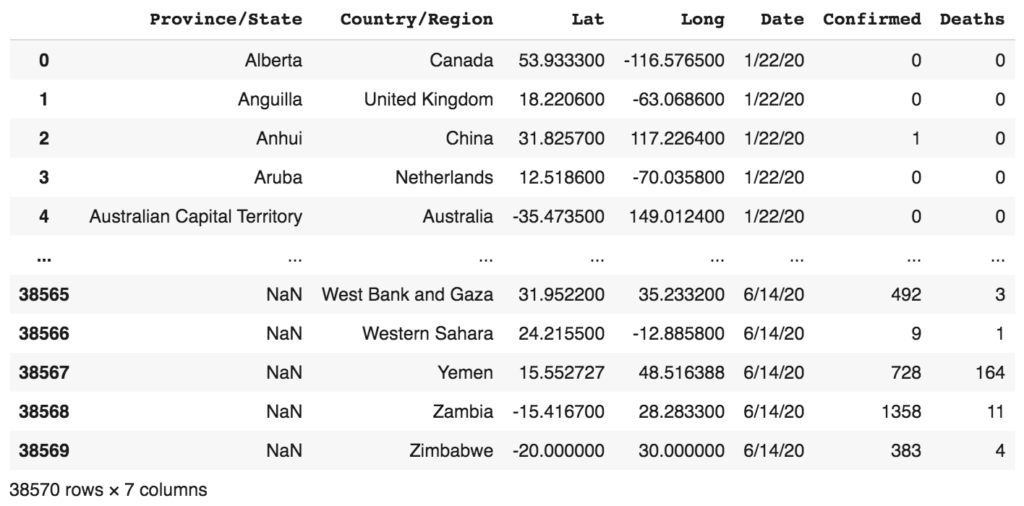
Je veux maintenant combiner ces DataFrames séparés en un seul afin qu’il soit plus facile d’effectuer des analyses par la suite :
df_combined = pd.concat([df_conf_melted,
df_death_melted["Deaths"]],
axis = 1,
sort = False)
df_combined
À ce stade, il serait utile de calculer le pourcentage de décès par rapport au nombre de cas confirmés :
df_combined["Percentage"] =
df_combined["Deaths"]/df_combined["Confirmed"]
df_combined
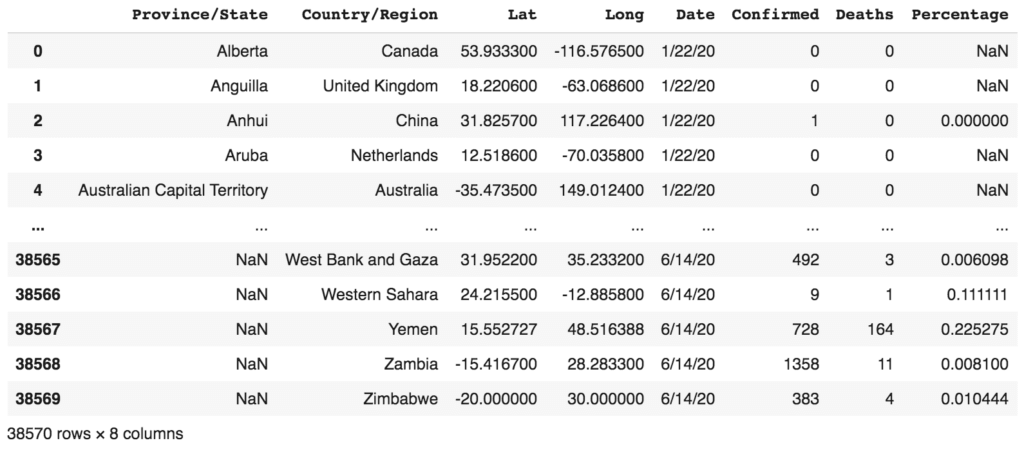
Parce qu’il y a des cellules qui sont des NaNs (causés par la division par zéro), il est important de les remplacer par des 0 :
df_combined.fillna(0, inplace=True) df_combined
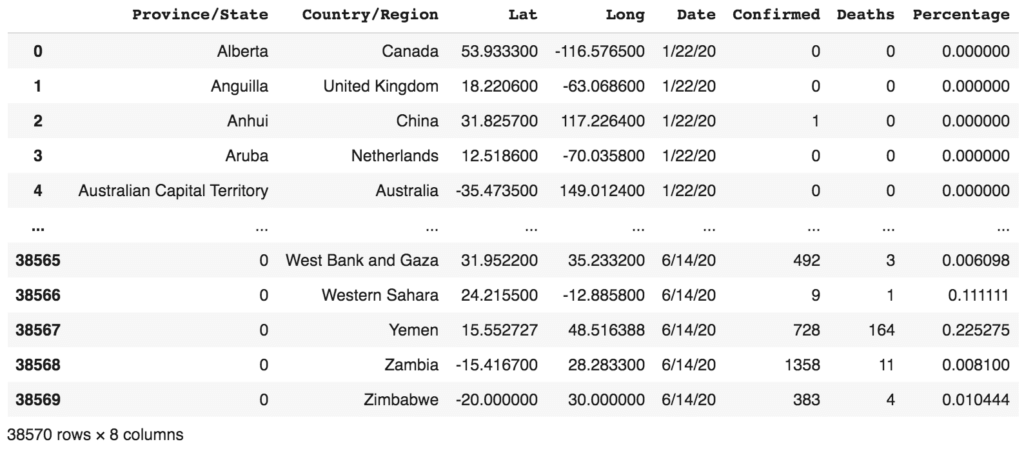
Il est utile de vérifier les types de données pour les différentes colonnes du DataFrame :
df_combined.dtypes ''' Province/State object Country/Region object Lat float64 Long float64 Date object Confirmed int64 Deaths int64 Percentage float64 '''
En particulier, la colonne Date est de type Objet (i.e. String).
Convertir la colonne Date en format datetime
Nous devons convertir la colonne Date en un format datetime.date afin de pouvoir effectuer plus tard un tri basé sur les dates.
import datetime
def format_time(datetime_str):
d = datetime.datetime.strptime(datetime_str, '%m/%d/%y')
return d.date()
df_combined["Date"] = df_combined["Date"].apply(format_time)
df_combined
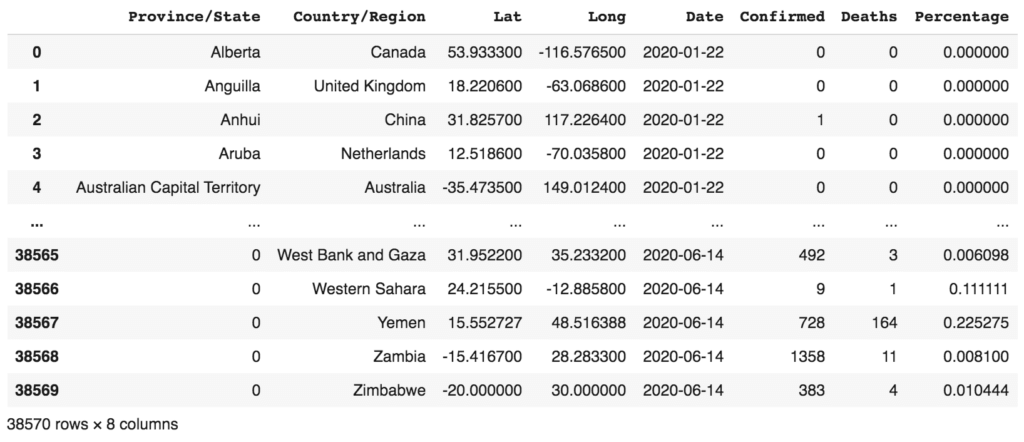
Afficher le nombre quotidien de cas confirmés et de décès
Maintenant que les DataFrames ont été combinés en un seul et considérablement nettoyés, nous pouvons commencer à effectuer des analyses afin de pouvoir en tirer des informations utiles.
Commençons par afficher le nombre quotidien de cas confirmés et de décès :
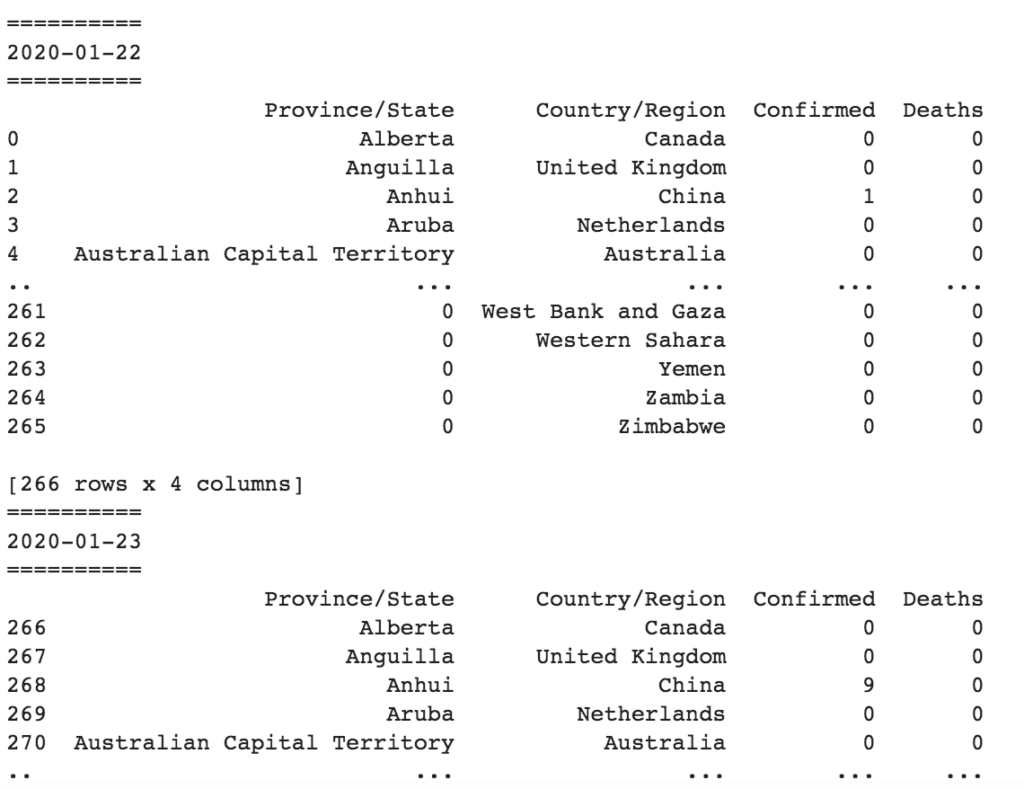
grouped_by_date = df_combined.groupby("Date")
for date, group in grouped_by_date:
print("==========")
print(date)
print("==========")
print(group[["Province/State","Country/Region", "Confirmed",
"Deaths"]])
Vous verrez maintenant le nombre de cas confirmés et de décès pour chaque jour (premier jour ci-dessous et début du second) :
Afficher le nombre total quotidien de décès confirmés et de décès pour chaque pays
Le résultat précédent indiquait le nombre pour chaque province/état dans chaque pays. Si vous souhaitez simplement connaître le nombre total pour chaque pays, vous pouvez effectuer une agrégation :
df_daily = df_combined.groupby(["Date","Country/Region"]).aggregate(
{'Confirmed': 'sum', 'Deaths': 'sum'})
df_daily
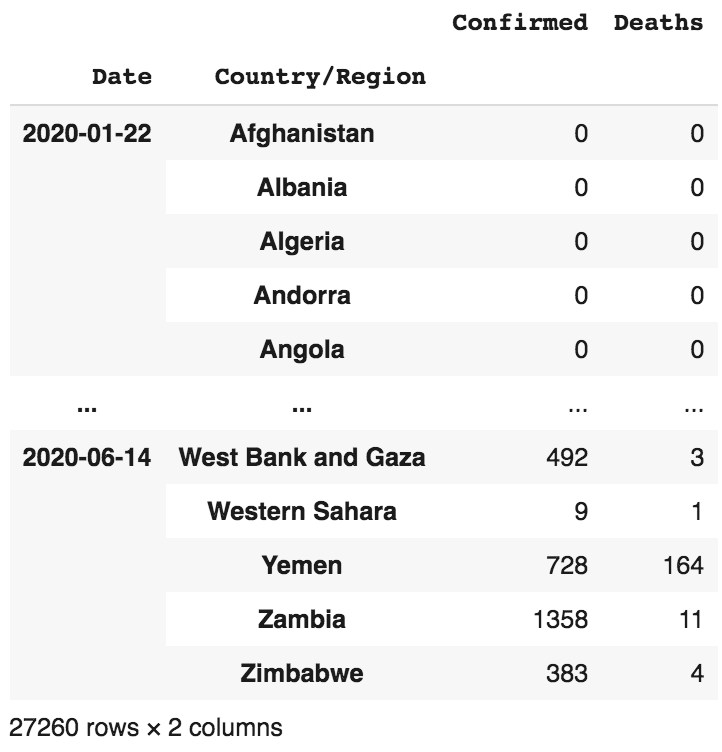
Obtenir les données pour un jour précis
Si vous voulez obtenir les chiffres pour un jour particulier, vous pouvez utiliser l’indexeur loc[] :
df_day = df_daily.loc[format_time('3/22/20')]
df_day
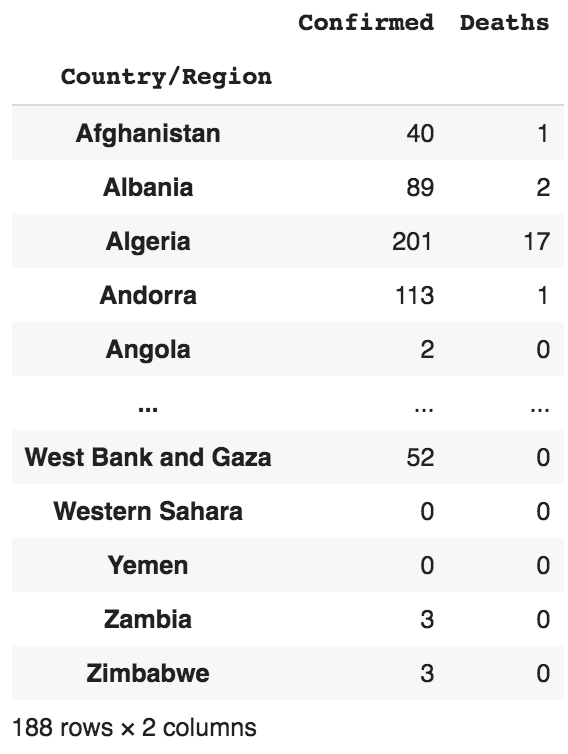
Obtenir les données pour un jour spécifique et pour un pays spécifique
Vous pouvez également récupérer les chiffres pour un pays particulier :
USA
df_day_usa = df_daily.loc[(format_time('5/22/20'),'US')]
df_day_usa
'''
Confirmed 1608623
Deaths 96296
Name: (2020-05-22, US), dtype: int64
'''
France
df_day_france = df_daily.loc[(format_time('5/22/20'),'France')]
df_day_france
'''
Confirmed 182354
Deaths 28292
Name: (2020-05-22, France), dtype: int64
'''
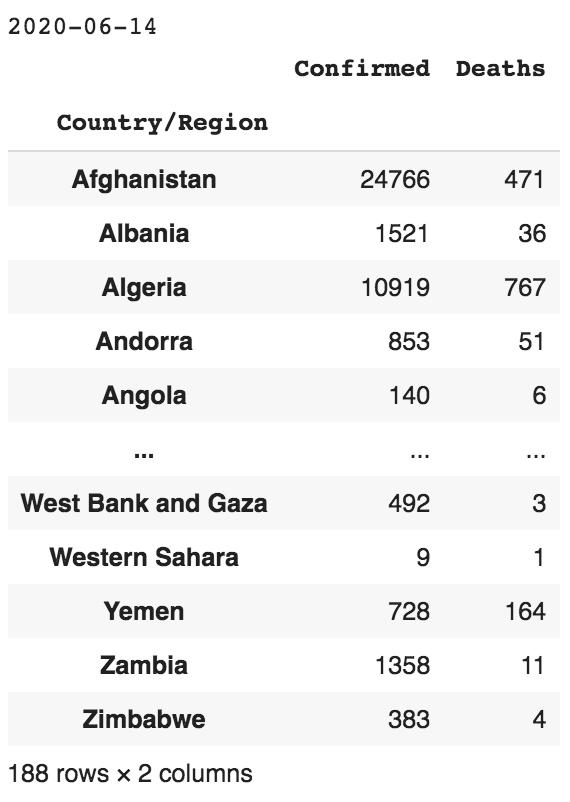
Afficher les données pour le jour le plus récent
Pour obtenir les chiffres du jour le plus récent, il faut d’abord obtenir la dernière date dans le DataFrame, puis l’utiliser pour extraire les données de cette date :
# Obtenir la date la plus récente most_recent_date = df_combined['Date'].max() print(most_recent_date) ''' 2020-06-14 ''' # Obtenir toutes les données pour la date la plus récente df_most_recent = df_daily.loc[most_recent_date,:] df_most_recent
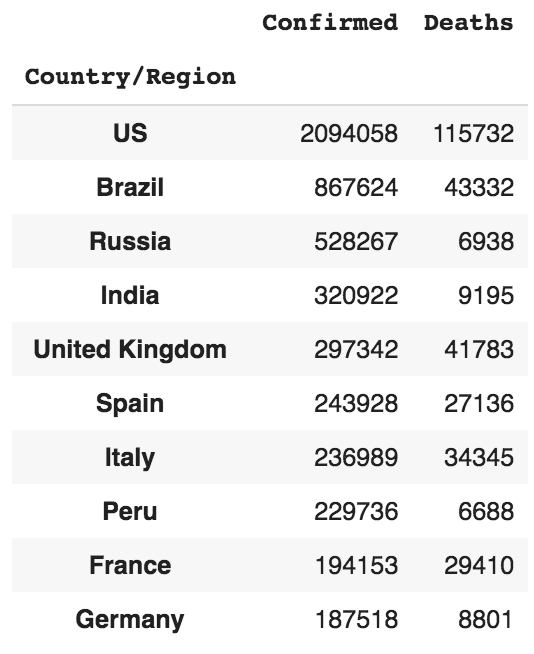
Les 10 premiers pays en terme de cas confirmés
Pour obtenir les 10 premiers pays avec des cas confirmés, triez le DataFrame df_most_recent par la colonne Confirmed et affichez ensuite les 10 premières lignes :
df_most_confirmed_recent_sorted = df_most_recent.sort_values(
by="Confirmed", ascending=False)
df_most_confirmed_recent_sorted.head(10)
Afficher les chiffres les plus récents pour un pays spécifique
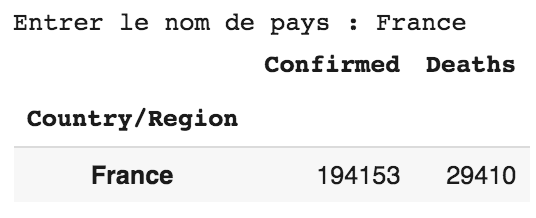
Vous pouvez utiliser la fonction input() pour demander à l’utilisateur d’entrer le pays dont il souhaite connaître les chiffres :
df_most_recent[df_most_recent.index == input("Entrer le nom de pays : ")]
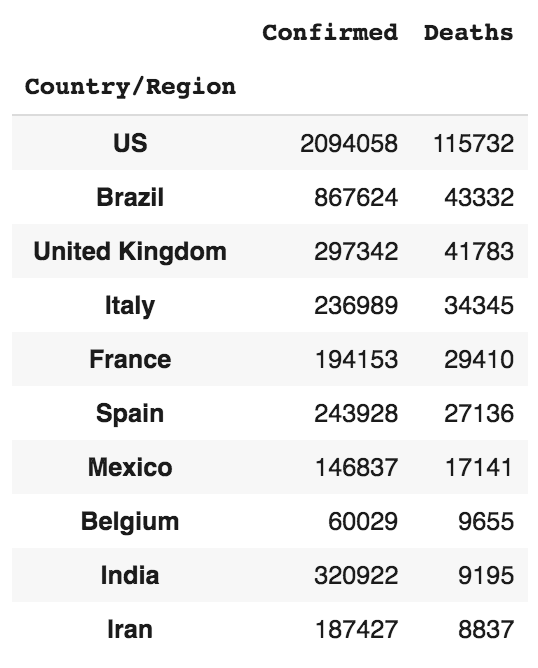
Les 10 pays où le plus grand nombre de décès ont été signalés
Pour obtenir les dix premiers pays où des décès ont été signalés, triez d’abord le DataFrame df_most_recent en utilisant la colonne Deaths. Ensuite, filtrez les pays qui n’ont pas signalé de décès. Enfin, affichez les 10 premières lignes :
df_most_deaths_recent_sorted = df_most_recent.sort_values(
by="Deaths", ascending=False)
df_most_deaths_recent_sorted = df_most_deaths_recent_sorted[df_most_deaths_recent_sorted.Deaths>0]
df_most_deaths_recent_sorted.head(10)
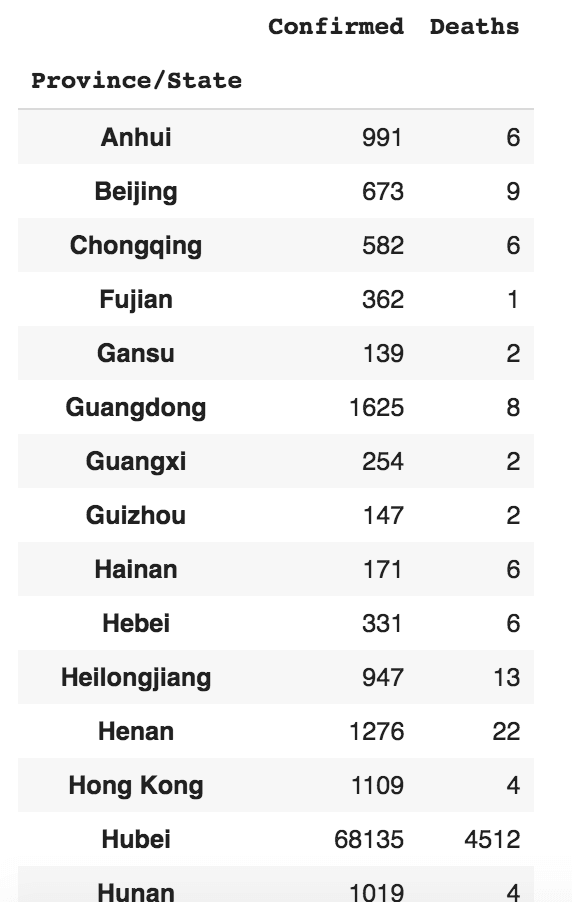
Afficher les données les plus récentes pour la Chine continentale
Dès les premiers jours de Covid-19, un certain nombre de provinces chinoises ont été infectées. Il serait donc intéressant de voir les chiffres des différentes provinces chinoises :
# obtenir toutes les lignes pour la Chine continentale
df_combined_china = df_combined[df_combined['Country/Region'] == "China"]
# en utilisant le dataframe de la chine, grouper par date et par province
df_combined_china_date = df_combined_china.groupby(["Date","Province/State"]).aggregate(
{'Confirmed': 'sum', 'Deaths': 'sum'})
# trouver les données pour la date la plus récente
df_combined_china_most_recent = df_combined_china_date.loc[most_recent_date,:]
df_combined_china_most_recent
Afficher les 10 premières provinces de Chine avec des cas confirmés
Pour obtenir les dix premières provinces de Chine présentant le plus grand nombre de cas confirmés, triez le DataFrame df_combined_china_most_recent par la colonne Confirmed et obtenez ses dix premières lignes :
df_combined_china_most_recent_sorted = df_combined_china_most_recent.sort_values(
by="Confirmed", ascending=False).head(10)
df_combined_china_most_recent_sorted
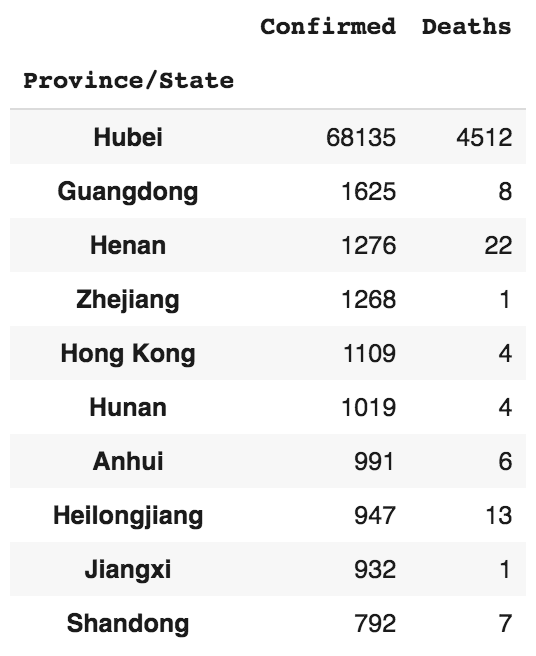
Comme vous pouvez le constater, Hubei est la province la plus touchée en Chine.
Le foyer du Covid-19 a été identifié pour la première fois à Wuhan (Wuhan est la capitale de la province de Hubei en République populaire de Chine).
Conclusion
J’espère que vous trouverez cet article utile et qu’il vous aidera à vous lancer dans l’analyse de données en Python.
Dans le prochain article, je vous montrerai comment utiliser la bibliothèque matplotlib pour effectuer une visualisation sur l’ensemble de données Covid-19.
La deuxième partie de cet article est disponible en cliquant ici.
Vous trouverez en cliquant ici tout le code de l’analyse de données sur le Covid-19 sur ce notebook Google Colab.