Nous savons tous déjà que Pandas et NumPy sont des bibliothèques étonnantes, et qu’elles jouent un rôle crucial dans nos analyses de données quotidiennes. Sans Pandas et NumPy, nous serions un peu perdus dans ce vaste monde de la Data Science. Aujourd’hui, je vais vous faire découvrir 12 fonctions Pandas et NumPy pour la Data Science qui vous faciliteront la vie et l’analyse.
Commençons par NumPy.
Fonctions NumPy
NumPy est le paquet fondamental pour le calcul scientifique avec Python. Il contient entre autres des choses comme :
- un puissant objet de type tableau à N dimensions
- des fonctions (de broadcasting) sophistiquées
- des outils pour l’intégration du code C/C++ et Fortran
- des capacités utiles d’algèbre linéaire, de transformée de Fourier et de nombres aléatoires
Outre ses utilisations scientifiques évidentes, NumPy peut également être utilisé comme un conteneur multidimensionnel efficace de données génériques. Des types de données arbitraires peuvent être définis. Cela permet à NumPy de s’intégrer de manière transparente et rapide à une grande variété de bases de données.
1. argpartition()
NumPy possède cette fonction étonnante qui permet de trouver l’indice des N plus grandes valeurs. La sortie sera l’indice des N plus grandes valeurs, et nous pourrons alors trier les valeurs si nécessaire.
# Tableau arbitraire x = np.array([12, 10, 12, 0, 6, 8, 9, 1, 16, 4, 6, 0]) index_val = np.argpartition(x, -4)[-4:] index_val # array([1, 8, 2, 0], dtype=int64) np.sort(x[index_val]) # array([10, 12, 12, 16])
2. allclose()
Allclose() est utilisé pour faire correspondre deux tableaux et obtenir la sortie en termes de valeur booléenne. Elle renvoie False si les éléments de deux tableaux ne sont pas égaux avec une certaine tolérance. C’est un excellent moyen de vérifier si deux tableaux sont similaires, ce qui peut en fait être difficile à mettre en œuvre manuellement.
array1 = np.array([0.12,0.17,0.24,0.29]) array2 = np.array([0.13,0.19,0.26,0.31]) # avec une tolérance de 0,1, il devrait retourner False : np.allclose(array1,array2,0.1) # False # avec une tolérance de 0,2, il devrait retourner True : np.allclose(array1,array2,0.2) # True
3. clip()
Clip() est utilisé pour conserver les valeurs d’un tableau dans un intervalle précis de valeurs. Parfois, nous devons garder les valeurs dans une limite supérieure et inférieure. Pour ce faire, nous pouvons utiliser la fonction clip() de NumPy. Lorsque l’on donne un intervalle, les valeurs en dehors de l’intervalle sont coupées sur les bords de l’intervalle.
x = np.array([3, 17, 14, 23, 2, 2, 6, 8, 1, 2, 16, 0]) np.clip(x,2,5) # array([3, 5, 5, 5, 2, 2, 5, 5, 2, 2, 5, 2])
4. extract()
Extract() comme son nom l’indique, est utilisé pour extraire des éléments spécifiques d’un tableau basé sur une certaine condition. Avec extract(), on peut aussi utiliser des conditions comme and et or.
# Nombres entiers aléatoires
array = np.random.randint(20, size=12)
array
# array([17, 14, 6, 10, 12, 4, 13, 4, 3, 11, 0, 10])
# Diviser par 2 et vérifier si le reste est égal à 1
cond = np.mod(array, 2)==1
cond
# array([ True, False, False, False, False, False, True, False, True,
True, False, False])
# Utiliser extract() pour obtenir les valeurs
np.extract(cond, array)
# array([17, 13, 3, 11])
# Appliquer directement la condition sur extract()
np.extract(((array < 3) | (array > 15)), array)
# array([17, 0])5. where()
Where() est utilisé pour renvoyer les éléments d’un tableau qui satisfont une certaine condition. Il renvoie la position de l’index des valeurs qui répondent à une certaine condition. C’est presque similaire à la condition where que nous utilisons en SQL, je vais le démontrer dans les exemples ci-dessous.
y = np.array([1,5,6,8,1,7,3,6,9]) # Lorsque y est supérieur à 5, renvoie la position de l'indice np.where(y>5) # array([2, 3, 5, 7, 8], dtype=int64),) # On remplace d'abord les valeurs qui correspondent à la condition, # ensuite on remplace les valeurs qui ne correspondent pas np.where(y>5, "OK", "NON") # array(['NON', 'NON', 'OK', 'OK', 'NON', 'OK', 'NON', 'OK', 'OK'],dtype='<U4')
6. percentile()
Percentile() est utilisé pour calculer le n-ième percentile des éléments du tableau le long de l’axe spécifié.
a = np.array([1,5,6,8,1,7,3,6,9])
print("50-ième Percentile de, axis = 0 : ",
np.percentile(a, 50, axis =0))
50-ème Percentile de, axis = 0 : 6.0
b = np.array([[10, 7, 4], [3, 2, 1]])
print("30-ième Percentile de b, axis = 0 : ",
np.percentile(b, 30, axis =0))
30-ième Percentile de b, axis = 0 : [5.1 3.5 1.9]Dites moi si vous les avez déjà utilisés et dans quelle mesure ils vous ont aidé. Passons maintenant aux fonctions incroyables de Pandas.
Fonctions Pandas
Pandas est un paquet Python fournissant des structures de données rapides, flexibles et expressives conçues pour rendre le travail avec des données structurées (tabulaires, multidimensionnelles, potentiellement hétérogènes) et des séries temporelles, à la fois facile et intuitif.
Pandas est bien adapté à de nombreux types de données différentes :
- Données tabulaires avec des colonnes de type hétérogène, comme dans un tableau SQL ou une feuille de calcul Excel
- Données de séries temporelles ordonnées et non ordonnées (pas nécessairement à fréquence fixe).
- Données matricielles arbitraires (typographiées de manière homogène ou hétérogène) avec des étiquettes de lignes et de colonnes
- Toute autre forme d’ensembles de données d’observations/statistiques. Il n’est en fait pas du tout nécessaire d’étiqueter les données pour les placer dans une structure de données Pandas.
Voici quelques-unes des choses que Pandas fait très bien :
- Manipulation aisée des données manquantes (représentées sous forme de NaN) en virgule flottante ainsi que des données en virgule non flottante
- Mutabilité de la taille : les colonnes peuvent être insérées et supprimées depuis un DataFrame et depuis tout objet de dimensions supérieures
- Alignement automatique et explicite des données : les objets peuvent être explicitement alignés sur un ensemble d’étiquettes (labels), ou l’utilisateur peut simplement ignorer les étiquettes et laisser les objets Series, DataFrame, etc. aligner automatiquement les données pour lui dans les calculs
- Fonctionnalité puissante et flexible de groupement en effectuant des opérations de split-apply-combine sur des ensembles de données, à la fois pour l’agrégation et la transformation de données
- Faciliter la conversion de données en désordre, différemment indexées dans d’autres structures de données (comme Python et NumPy) en objets DataFrame
- Découpage intelligent basé sur l’étiquette, indexation fantaisiste et sous-ensemble de grands ensembles de données
- Fusion et assemblage intuitifs d’ensembles de données
- Remodelage et pivotement flexibles des ensembles de données
- Étiquetage hiérarchique des axes (possibilité d’avoir plusieurs étiquettes par marqueur)
- Des outils d’I-O (Input-Output) robustes pour le chargement de données à partir de fichiers (CSV et autres délimités), de fichiers Excel, de bases de données, et pour la sauvegarde/le chargement de données à partir du format ultra-rapide HDF5
- Fonctionnalités spécifiques aux séries temporelles : génération de plages de dates et conversion de fréquences, statistiques de fenêtres mobiles, décalage et retard de dates.
7. read_csv(nrows=n)
Vous connaissez peut-être déjà l’utilisation de la fonction read_csv. Mais la plupart d’entre nous commettent encore l’erreur de lire le fichier .csv en entier même si cela n’est pas nécessaire. Considérons une situation où nous ne connaissons pas les colonnes et les données présentes dans un fichier .csv de 10 Go. La lecture du fichier .csv en entier ne serait pas une décision intelligente, car elle constituerait une utilisation inutile de notre mémoire et prendrait beaucoup de temps. Nous pouvons simplement importer quelques lignes du fichier .csv et poursuivre la lecture selon nos besoins.
import io
import requests
# J'utilise cet ensemble de données en ligne juste pour vous faciliter la tâche
url = "https://raw.githubusercontent.com/moncoachdata/Datasets/master/AirPassengers.csv"
s = requests.get(url).content
# ne lire que les 10 premières lignes
df = pd.read_csv(io.StringIO(s.decode('utf-8')), nrows=10 , index_col=0)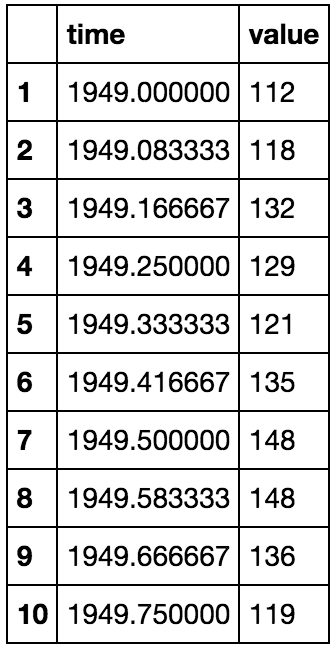
8. map()
La fonction map() est utilisée pour cartographier les valeurs des séries en fonction de la correspondance d’entrée. Elle est utilisée pour substituer chaque valeur d’une série par une autre valeur, qui peut être dérivée d’une fonction, d’un dictionnaire ou d’une série.
# créer un DataFrame
dframe = pd.DataFrame(np.random.randn(4, 3), columns=list('bde'), index=['India', 'USA', 'China', 'Russia'])
dframe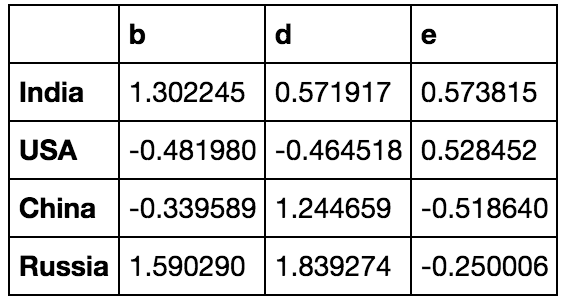
# calculer une chaîne formatée à partir de chaque valeur en virgule flottante dans le tableau changefn = lambda x: '%.2f' % x # Apporter des changements au niveau des éléments dframe['d'].map(changefn) ''' India 0.57 USA -0.46 China 1.24 Russia 1.84 Name: d, dtype: object '''
9. apply()
La méthode apply() permet aux utilisateurs de passer une fonction et de l’appliquer sur chaque valeur de la série Pandas.
# max minus mix lambda fn fn = lambda x: x.max() - x.min() # Appliquez ceci sur le DataFrame que nous venons de créer ci-dessus dframe.apply(fn) ''' b 2.072270 d 2.303792 e 1.092456 dtype: float64 '''
10. isin()
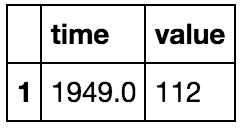
La méthode isin() est utilisé pour filtrer les DataFrames. isin() aide à sélectionner les lignes ayant une valeur particulière (ou multiple) dans une colonne particulière. C’est la fonction la plus utile que j’ai rencontrée.
# En utilisant le DataFrame que nous avons créé pour read_csv filter1 = df["value"].isin([112]) filter2 = df["time"].isin([1949.000000]) df [filter1 & filter2]
11. copy()
La fonction copy() est utilisée pour créer une copie d’un objet Pandas. Lorsque vous affectez un DataFrame à un DataFrame, sa valeur change lorsque vous effectuez des modifications dans l’autre. Pour éviter ce problème, nous pouvons utiliser la fonction copy().
# créer une série d'échantillons data = pd.Series(['India', 'Pakistan', 'China', 'Mongolia']) # Affectation des problèmes auxquels nous sommes confrontés data1= data # Changer une valeur data1[0]='USA' # Change également la valeur dans l'ancien DataFrame data ''' 0 USA 1 Pakistan 2 China 3 Mongolia dtype: object ''' # Pour éviter cela, nous utilisons la création de copies de séries new = data.copy() # attribution de nouvelles valeurs new[1]='Valeur changée' # impression des données print(new) print(data) ''' 0 India 1 Valeur changée 2 China 3 Mongolia dtype: object 0 India 1 Pakistan 2 China 3 Mongolia dtype: object '''
12. select_dtypes()
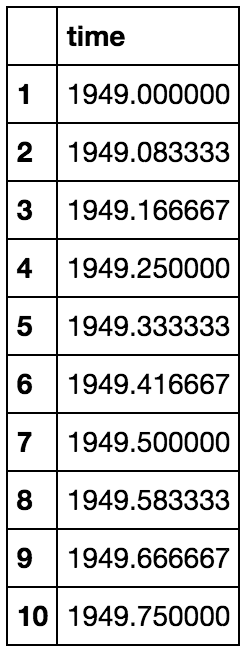
La fonction select_dtypes() renvoie un sous-ensemble des colonnes du DataFrame en fonction des dtypes des colonnes. Les paramètres de cette fonction peuvent être définis de manière à inclure toutes les colonnes ayant un type de données spécifique ou à exclure toutes les colonnes qui ont des types de données spécifiques.
# Nous utiliserons le même DataFrame que celle utilisée pour read_csv framex = df.select_dtypes(include="float64") # Retourne seulement la colonne time framex
Bonus : pivot_table()

La fonction la plus étonnante et la plus utile de Pandas est pivot_table. Si vous hésitez à utiliser groupby et souhaitez étendre ses fonctionnalités, vous pouvez très bien utiliser la fonction pivot_table. Si vous savez comment fonctionne le tableau croisé dynamique dans Excel, alors ce sera sans doute très simple pour vous. Les niveaux dans le tableau croisé dynamique seront stockés dans des objets MultiIndex (index hiérarchiques) sur l’index et les colonnes du DataFrame de résultat.
# Créer un DataFrame d'échantillons
school = pd.DataFrame({'A': ['Jay', 'Usher', 'Nicky', 'Romero', 'Will'],
'B': ['Masters', 'Graduate', 'Graduate', 'Masters', 'Graduate'],
'C': [26, 22, 20, 23, 24]})
# Créons un tableau croisé dynamique pour séparer les étudiants en fonction de leur âge et de leur parcours
table = pd.pivot_table(school, values ='A', index =['B', 'C'],
columns =['B'], aggfunc = np.sum, fill_value="Not Available")
table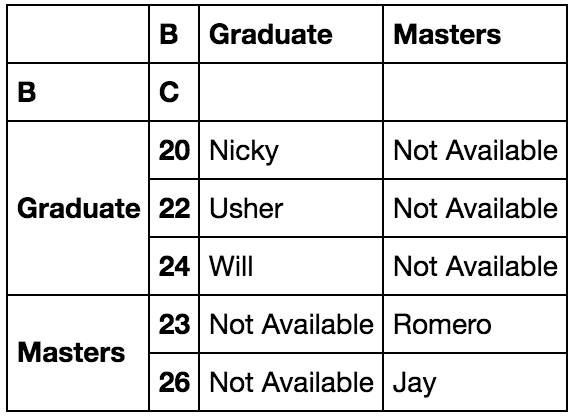
Faites-moi savoir ci-dessous dans les commentaires si vous avez rencontré ou utilisé d’autres fonctions étonnantes. J’aimerais en savoir plus à leur sujet 🙂
Lire aussi 10 astuces Pandas à connaître.